Decodering van het menselijk genoom: een mijlpaal binnen de natuurwetenschappen
In 1990 werd het internationale Human Genome Project (HGP) opgestart, met als einddoel: de decodering van het menselijk genoom. “Enkel de beste, met overheidsgelden gefinancierde laboratoria kunnen – ingebed in een internationaal overkoepelende structuur – een dergelijk titanenwerk tot een goed einde brengen”, aldus James Watson, laureaat voor de Nobelprijs én toenmalig projectleider (Figuur 1). En hij kreeg gelijk: de sequenering van het menselijk genoom zou uiteindelijk meer dan 10 jaar in beslag nemen en zou allicht nog langer geduurd hebben als Craig Venter, toenmalig CEO van het biotech bedrijf Celera Genomics, zich niet in de strijd had gemengd en in 1998 een gelijkaardig project opgestart had. In 2000 werd tijdens een gemeenschappelijke persconferentie – tot stand gekomen onder niet geringe politieke druk – in het Witte Huis een eerste draft voorgesteld van het menselijk genoom. De volledige DNA-sequentie (3 miljard basenparen) van Homo sapiens sapiens zou uiteindelijk twee jaar later gepubliceerd worden. Kostprijs: 3 miljard US$.
Sindsdien heeft de sequentie -en informaticatechnologie een explosieve ontwikkeling doorgemaakt. Begin dit jaar kondigde het biotechnologiebedrijf Illumina Inc. aan het volledige menselijke genoom te decoderen binnen de 24 uur, en dat voor een fractie van de oorspronkelijke kostprijs: 1000 US$ (insiders verwachten dat deze prijs de volgende jaren zal dalen tot 200 US$). Dit betekent dat in een niet al te verre toekomst de sequenering van het individuele genoom binnen het bereik van de ‘gewone man’ zal komen.
| Figuur 1: James Watson en Francis Crick. In 1962 zullen ze, samen met Maurice Wilkins, voor de opheldering van de DNA structuur de nobelprijs voor geneeskunde of fysiologie ontvangen. |
|---|
 |
We kennen de volgorde van de letters, maar kunnen we ook het boek lezen?
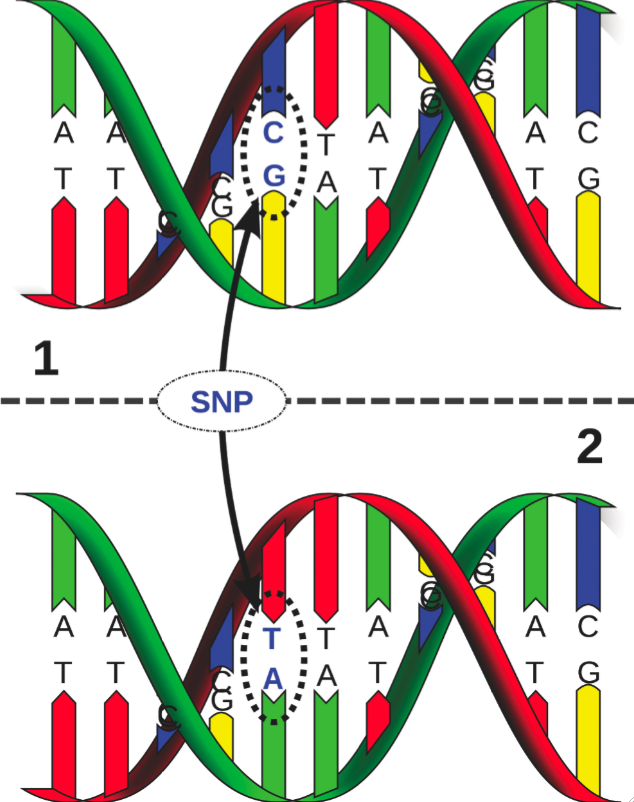
Inmiddels zijn de (ongeveer) 25.000 genen die in ons genoom vervat liggen bekend – overigens veel minder dan de meeste specialisten een decennium geleden hadden voorspeld. Sindsdien vormt het ophelderen van hun functie(s) het onderwerp van intensief wetenschappelijk onderzoek. Klinisch belangrijk binnen deze context zijn de zgn. enkel-nucleotide polymorfismen (Single Nucleotide Polymorphisms, kortweg SNP’s): individuele variaties van één enkel nucleotide in ons DNA (Figuur 2). Ongeveer 90% van alle genetische variaties binnen het menselijk genoom bestaat uit SNP-polymorfismen: zij vormen de genetische basis van onze identiteit. De contributie van een SNP is vaak onduidelijk – de wijziging van bepaalde nucleotiden in ons DNA levert ogenschijnlijk geen evolutief voordeel of nadeel op – maar in bepaalde gevallen zijn zij significant geassocieerd met een pathologie. De jongste jaren is het academisch en klinisch geörienteerde onderzoek naar het opsporen van SNP’s en hun relatie tot bepaalde aandoeningen explosief toegenomen. Onderzoekers en clinici verwachten dat dergelijke research onder meer zal bijdragen tot meer gepersonaliseerde behandelingswijzen in tal van medische domeinen. Gevraagd naar zijn mening hieromtrent antwoordde de Amerikaanse geneticus Francis Collins, opvolger van James Watson als HGP projectleider: “Voor sommige aandoeningen kunnen we aan de hand van SNP’s reeds vrij accuraat voorspellen welke personen een verhoogd risico lopen. Laat ons evenwel duidelijk zijn: we staan aan het prille begin van een ontwikkeling en de ingrijpende klinische benefits van genoomanalyses ligt vóór ons.”
| Figuur 2: SNP’s (enkel-nucleotide polymorfismen) of combinaties van SNP’s zijn in sommige gevallen significant geassocieerd met een aandoening. |
|---|
 |
Genoom analyse via het internet
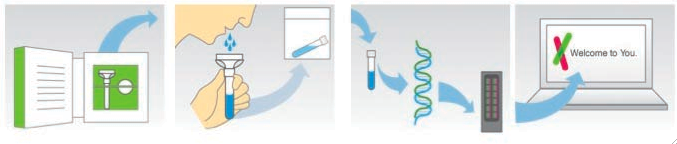
Een aantal Amerikaanse firma’s (o.a. 23andMe en deCODEme) bieden via het internet een persoonlijke genoomanalyse aan voor minder dan 100 US$. Via hun website (www.23andme.com, www.decodeme.com) kan een kit besteld worden, waarmee een speekselstaal ontnomen kan worden, dat nadien wordt opgestuurd (Figuur 3). Gebruik makend van (door Illumina Inc. ontwikkelde) DNA microarray-technologie, wordt het DNA uit het staal geanalyseerd op de aanwezigheid van 1 miljoen SNP’s. Van de betreffende persoon wordt m.a.w. een SNP-profiel opgemaakt (weliswaar nog onvolledig, aangezien schattingen ervan uitgaan dat ongeveer 10 miljoen SNP’s verspreid liggen over het menselijk genoom). Via de website kan de interpretatie van de genetische data een week later opgevraagd worden. Het individuele genetische profiel verschaft informatie over:
-
genetische aandoeningen, zoals chorea huntington of mucoviscidose. De genoomanalyse geeft uitsluitsel of de persoon drager is van recessieve (ziektegerelateerde) allele;
-
de individuele respons tegenover bepaalde therapieën. Een gekend voorbeeld in dit verband is het anticoagulans warfarine, dat slechts werkzaam is binnen een nauw therapeutisch venster en bij sommige patiënten interne en fatale bloedingen tot gevolg kan hebben. De werkzaamheid van warfarine wordt (naast andere parameters zoals bvb. de ouderdom en de lichaamsbouw van de persoon) voor 40% bepaald door twee genen: cytochrome P-450 reductase (CYP2C9) en vitamine K epoxide reductase (VKORC1). Door het bepalen van de genetische SNP-variant kan de individuele respons tegenover het anticoagulans predictief bepaald worden en is de werkzame, veilige dosis van warfarine bekend. Een ander voorbeeld is de selectieve oestrogeenreceptormodulator tamoxifen, dat ingezet wordt ter behandeling van bepaalde vormen van borstkanker. Het geneesmiddel vertoont geen therapeutische werking bij ongeveer 6% van de patiënten, als gevolg van een polymorfisme in het cytochroom P450 3A4 gen (CYP3A4). Een genoomanalyse kan uitsluitsel geven tot welke subpopulatie men behoort;
-
het risicoprofiel voor een aantal aandoeningen zoals type 2 diabetes, borstkanker, colonkanker, cardiovasculaire en cerebrovasculaire aandoeningen, ouderdomgerelateerde maculadegeneratie en de ziekte van Alzheimer.
| Figuur 3: Genoomanalyse via het internet. Men bestelt een kit, waarmee een speekselstaal ontnomen wordt. Het DNA uit dit staal wordt geanalyseerd op de aanwezigheid van 1 miljoen SNP’s. Eén week later kan men zijn risicoprofiel voor een aantal aandoeningen opvragen. |
|---|
 |
Het zal eenieder duidelijk zijn: individuele genoomanalyses kunnen bijdragen tot het ontwikkelen van meer gepersonaliseerde therapieën doordat de genetische data van een persoon (kunnen) ingebed worden in zijn gezondheidsprofiel. Maar gebeurt dit wel? Niet weinig specialisten menen dat deze bio-informaticageneeskunde de DNA-analyse naar het middelpunt van de diagnosestelling én therapeutische behandeling rukt zonder rekening te houden met alle andere klinisch relevante parameters.
Risicoprofiel per muisclick?
Een centrale vraag binnen dit discours is: hoe geloofwaardig zijn genoomanalyses die via het internet aangeboden worden? Is het inderdaad mogelijk om aan de hand van een bepaalde combinatie van SNP’s een betrouwbaar risicoprofiel op te stellen voor een gegeven aandoening? De meningen hierover zijn verdeeld. Vele aandoeningen zijn immers multifactorieel, en niet alleen de genetische component (als die al bestaat), maar ook omgevingsfactoren en levensstijl zijn bepalend voor het al dan niet optreden van een ziekte. Een typisch voorbeeld is type 2 diabetes, een aandoening die – naast een genetische predispositie – bepaald wordt door klinische parameters die sterk gerelateerd zijn aan leefgewoonten, zoals obesitas, voedingspatroon, alcoholconsumptie en een gebrek aan beweging. Inderdaad, de predictieve waarde van deze parameters (die niet uit een DNA-analyse af te lezen zijn) om het risico op type 2 diabetes in te schatten, is op dit ogenblik accurater dan een gegeven combinatie van SNP’s. Hetzelfde geldt voor vele cardiovasculaire aandoeningen en tal van andere ziekten. Om het eenvoudig te stellen: de relatie tussen onze genetische fingerprint en omgevingsfactoren is bepalend voor ons gezondheidsprofiel. Een risicoprognose louter gebaseerd op een DNA-analyse is volgens vele specialisten dan ook weinig significant.
Prof. Em. Uta Francke (Genetics & Pediatrics Dept., Stanford Medical School en Senior Medical Director bij 23andMe) betwist geenszins dat de risicoinschattingen voor heel wat aandoeningen op dit ogenblik nog vaag zijn. “Maar”, zo meent Uta Francke: “voor een aantal aandoeningen kan wél reeds een aanvaardbaar risicoprofiel geformuleerd worden.” (23andMe stelt op dit ogenblik een risicoprofiel op voor 97 aandoeningen, deCODEme doet hetzelfde voor 47 aandoeningen). Zij benadrukt tevens dat de prognoses in de toekomst steeds accurater zullen worden, en wel om de volgende redenen. Ten eerste bevat de databank van 23andMe op dit ogenblik ongeveer 125.000 individuele genoomanalyses. Dit aantal neemt elk dag toe. Deze data worden voortdurend getoetst aan de meest up-to-date biomedische literatuur, en laten aldus toe om nieuwe associaties tussen genotypering (zoals SNP combinaties) en bepaalde risicoprofielen bloot te leggen. Naarmate meer data gecollecteerd worden, zullen de predicties nauwkeuriger worden. De geteste personen kunnen trouwens regelmatig een update van hun risicoprofiel opvragen via de website. Ten tweede voert 23andMe bij de geteste personen internetbevragingen door om informatie te bekomen over o.a. hun levensstijl, lichaamsbouw, eetgewoonten, familiale aandoeningen en hun algemene gezondheidstoestand. Hiermee willen ze een zo nauwkeurig mogelijk beeld verkrijgen van alle klinisch relevante parameters. Meer dan 70% van de geteste personen hebben deze bevragingen ingevuld en zich ermee akkoord verklaard de data (anoniem) voor wetenschappelijke doeleinden aan te wenden. Dat gebeurt dan ook: vorig jaar verscheen o.a. een publicatie van het 23andMe wetenschappelijk team, waarbij twee nieuwe loci geïdentificeerd werden die significant met de ziekte van Parkinson geassocieerd zijn (Do C.B. et al., PloS Genetics, Juni 2011).
Farmaceutische bedrijven zijn geïnteresseerd
Uta Francke benadrukt overigens dat het biotechnologie bedrijf 23andMe een samenwerking met de farmaceutische industrie in de toekomst niet uitsluit. Bij het ontwikkelen van bepaalde geneesmiddelen speelt de genetische diagnostiek immers een niet geringe rol en is ze mede bepalend voor het welslagen van een therapie (voorbeelden: cetuximab ter behandeling van colorectale kanker, trastuzumab t.b.v. borstkanker en imatinib t.b.v. chronische myeloïde leukemie). Een aantal farmaceutische bedrijven zijn dan ook sterk geïnteresseerd in individuele DNA-profielen, die – en dit is belangrijk – representatief zijn voor een grote, at random-populatie.
Hoe gaan we met de gegevens om?
Bij een genoomanalyse, zoals die door 23andMe en deCODEme aangeboden wordt, verneemt een persoon via het internet zijn risicopercentage voor deze of gene ziekte. Deze gegevens – let wel: het gaat hier in een aantal gevallen om ernstige, deels levensbedreigende aandoeningen – worden evenwel op generlei wijze geïnterpreteerd door een vakpersoon of een behandelende arts. Het is duidelijk dat dit in bepaalde gevallen (bvb. bij een sterk verhoogd risico voor kanker) bij sommige personen angsten en stress kan uitlokken. Deze prognoses kunnen bovendien mensen beïnvloeden bij een aantal belangrijke keuzes in hun leven. Kijkt iemand die geïdentificeerd werd als drager van een mutant gen voor een ernstige, congenitale afwijking – nogmaals: zonder enige medische omkadering – anders aan tegen het huwelijk of het op de wereld zetten van kinderen? Zal iemand met een sterk verhoogd risico op de ziekte van Alzheimer (waarvoor nog steeds geen adequate behandeling werd ontwikkeld) dit ervaren als een uitgesteld doodvonnis? Een aantal studies in de Verenigde Staten – waar deze discussie op dit ogenblik gevoerd wordt – proberen hieromtrent inzicht te verschaffen. Het gevaar lijkt inderdaad niet denkbeeldig dat een aantal personen hun risicoprofiel over-interpreteren en verwarren met een prognose voor de toekomst. Anderzijds kan een confrontatie met het persoonlijke riscoprofiel ook voordeel bieden. Francis Collins vermeldt in een recent interview dat zijn DNA-analyse een verhoogd risico had aangetoond voor type 2 diabetes. “Ik werd wakker geschud”, aldus Collins: “ik heb mijn voedingspatroon gewijzigd en ben rigoureus en gedisciplineerd gaan sporten. Ik heb opnieuw mijn optimaal gewicht en voel me beter dan ooit tevoren.” Men kan zich effectief scenario’s voorstellen waarbij iemand met bijvoorbeeld een verhoogd risico op colonkanker regelmatig een colonoscopie laat uitvoeren en preventief poliepen laat verwijderen alvorens deze (mogelijks) in een maligne richting evolueren.
Uitdagingen voor de toekomst
In medische kringen wordt algemeen aangenomen dat de diagnostiek en de therapeutische behandeling in de toekomst op een meer gepersonaliseerde wijze zullen doorgevoerd worden. Genetische tests – die in bepaalde gevallen reeds worden aangewend – zullen in een aantal klinische domeinen aan belang winnen bij de eenduidige karakterisatie van een patiënt. Weinigen betwijfelen dat ook genoomanalyses (volledige DNA-seqenering of karakterisering van SNP’s) in bepaalde gevallen een bijdrage kunnen leveren tot het stellen van een diagnose of het bepalen van de optimale therapie. Sommige experts zijn echter van mening dat genoomanalyses routinematig zullen doorgevoerd worden ter bepaling van het individuele gezondheids- én risicoprofiel. Uta Francke is in ieder geval duidelijk: “In de ontwikkelde landen zullen in de toekomst de meeste personen hun volledige genoomsequentie kennen, daarvan ben ik overtuigd. Misschien zal het zelfs zo zijn dat bij de geboorte van een kind een genoomsequentie routinematig doorgevoerd zal worden.” Men kan zich inderdaad voorstellen dat in de toekomst personen een chip met hun individuele DNA-sequentie bij zich dragen. Dit zou bijvoorbeeld de behandelende arts in een noodgeval toelaten om zeer snel de correcte therapeutische beslissing te nemen, bvb. bij het bepalen van de veilige, werkzame warfarinedosis.
Het is evenwel duidelijk dat een routinematig doorvoeren van genoomsequenering en de omgang met dergelijke medische informatie ethische en zelfs juridische vragen oproept. Het lijkt niet denkbeeldig dat de ziektekostenverzekering – met de steeds stijgende kosten in de gezondheidszorg –de resultaten van een genoomanalyse zal willen zien vooraleer een aangepaste verzekeringspolis op te maken. Zullen anderzijds artsen het deontologisch nog kunnen verantwoorden om zeer dure medische ingrepen door te voeren en/of exclusieve therapieën op te starten bij patiënten wiens DNA-analyse een nefast, en in de gegeven setting, klinisch relevant risicoprofiel aantoont? Het lijkt alleszins aannemelijk dat de resultaten van de genoomanalyse deel zullen uitmaken van het beslissingsproces. Wat indien aanstaande ouders om een prenatale genoomsequenering vragen? Op dit eigenste ogenblik vormt deze procedure op technologisch vlak immers geen onoverkomelijk probleem. Welke risicoprofielen zullen een afbreken van de zwangerschap (of het niet implanteren van een bevruchte eicel) rechtvaardigen? Welke mening men ook toegedaan is m.b.t. het populariseren van genoom analysen, de roep naar een omvattende ethische discussie en het scheppen van juridische duidelijkheid wordt in de Verenigde Staten almaar luider. Het lijkt enkel een kwestie van tijd alvorens dezelfde vragen de Atlantische Oceaan zullen overwaaien.